心情随笔
20230512_在单细胞水平上揭示基因的秘密:第三代测序的力量
454


目前缺少可以在单个细胞全基因组水平上检测结构变异(SVs)和染色体外的环状DNA(ecDNAs)的方法。为了解决这个问题,作者开发了一种基于第三代测序平台的单细胞全基因组测序方法,命名为SMOOTH-seq,并且作者评估了该方法在人类癌细胞系和结直肠癌样本中检测CNVs、SVs和SNVs的能力,并表明SMOOTH-seq能够可靠有效地检测单个细胞中的SVs和ecDNAs。
单细胞水平三代测序相较于传统的二代转录组测序和三代转录组测序的优势:
- 高分辨率:单细胞测序技术可以提供每一个细胞的基因表达信息,这是传统的基因测序技术无法实现的。这使得我们可以研究在不同细胞之间,甚至在同一种细胞的不同状态之间,基因表达的差异。这种高分辨率的信息对于我们理解生物体内的复杂生物学过程,例如发育、疾病发生和进展等,具有重要意义。
- 长读长:三代测序技术的一个显著特点就是能够产生长达数十千个碱基的读长(read length),这样可以直接得到完整的转录本,包括所有的外显子和内含子,以及其间的剪接位点,有助于我们更深入地理解基因的结构和功能。
- 更全面的信息:三代测序技术还可以检测到RNA分子的修饰,如甲基化等,这是二代测序技术无法做到的。这些修饰在基因表达调控、RNA稳定性、RNA结构形成等方面都起着重要作用,因此对于我们理解基因表达的调控机制具有重要意义。
- 降低扩增偏差:传统的二代测序需要通过PCR扩增来获取足够的DNA模板,而这个过程可能会引入偏差。而三代测序技术则可以直接对单个DNA或RNA分子进行测序,大大减少了扩增偏差。
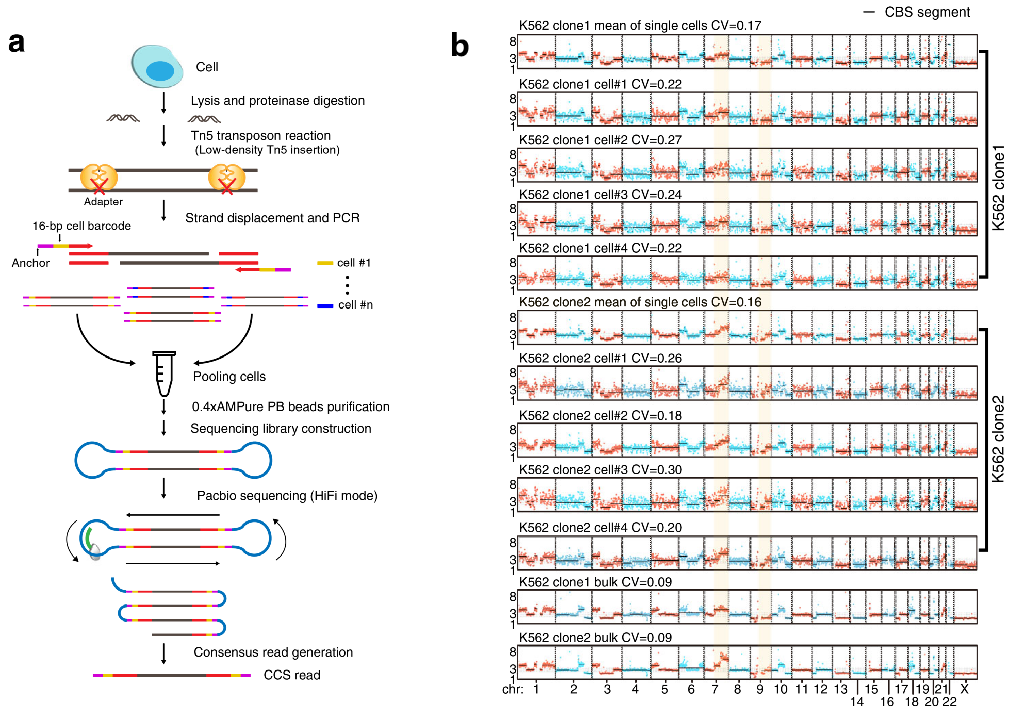
(a)Smooth-seq测序流程:
- DNA转座反应:首先,使用商业化的Tn5转座酶和特定的转座反应缓冲液,将待测细胞的基因组DNA(gDNA)与Tn5转座酶结合。这样,转座酶将在基因组DNA中随机插入适配子序列,形成带有适配子的转座片段。
- 长片段扩增:接下来,通过PCR反应扩增带有适配子的转座片段。在PCR反应中,使用Tks DNA聚合酶进行扩增,该酶适用于长片段DNA的扩增。PCR反应的循环数需要进行优化,以获得适当长度的扩增片段。
- 文库构建:扩增的长片段DNA经过纯化处理,常用的方法是使用磁珠(如AMPure PB beads)进行纯化。纯化后的长片段DNA作为文库构建的起点。使用特定的文库构建方法,将长片段DNA进行连接、修复和标记,以准备进行后续的测序。
- SMRT DNA测序:文库构建完成后,使用第三代测序平台(如SMRT DNA测序平台)对文库进行测序。这种测序方法产生的读段被称为环状连续一致序列(CCS reads),它们具有较长的读长和较高的准确性。CCS reads的长度可以达到几千到数万个碱基对,并且每个CCS read通常由多个亚读序列(subreads)组成。
(b)使用SMOOTH-seq检测单个K562细胞的拷贝数变异:
研究者利用SMOOTH-seq测序技术对K562细胞的拷贝数变异(CNV)进行了详细分析。CNV通常指的是长度大于1 kb的基因组片段的拷贝数增加或减少,这种变异往往是由基因组重排引发的。在这项研究中,研究者对两个K562细胞样本分别进行了单细胞测序和普通的转录组测序。在每个样本中,选取了四个单细胞进行测序,并将得到的序列与基因组进行比对,以确定每条染色体上的CNV。结果显示,每个样本内不同细胞的染色体上的CNV非常相似,而且与另一样本的单细胞测序结果也有高度相似性。这一发现验证了第三代单细胞测序技术可以有效用于CNV检测。变异系数(CV)是用来衡量数据分布情况的一个重要指标,计算公式为:CV = (标准偏差SD/平均值Mean)× 100%。图中前四个结果是样本一中四个单细胞的CNV检测结果,接下来的四个是样本二中四个单细胞的结果。最后两个结果是利用普通的基因组测序得到的CNV数据。可以观察到,每个细胞中,染色体上的CNV分布大部分是相同的,这验证了作者检测到的CNV数据的准确性。
使用SMOOTH-seq进行结构变异(SV)的检测
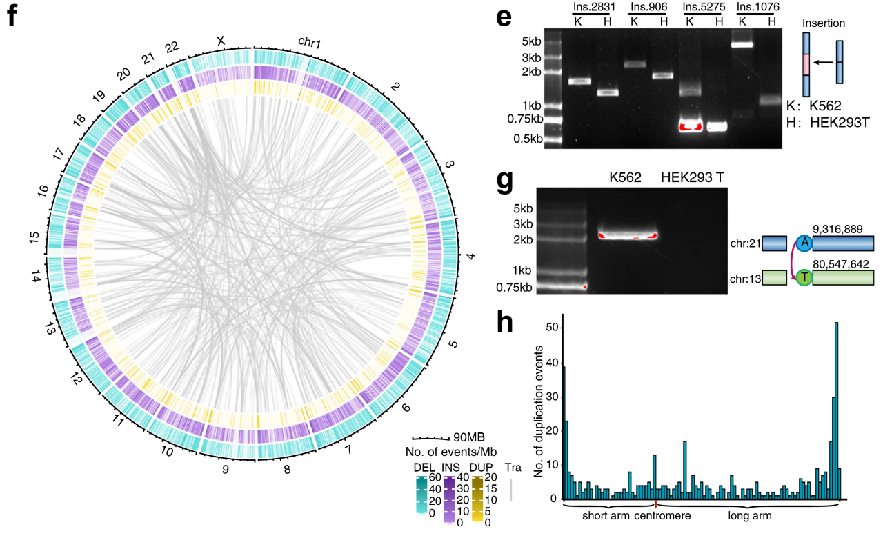
除了使用SMOOTH-seq检测拷贝数变异外,作者还尝试使用该方法来检测基因组结构变异,这些变异通常包括缺失、插入、重复、反转和易位。在下图a中,作者展示了在K562细胞的不同染色体上检测到的缺失、插入和重复突变情况。蓝色区域表示发生缺失突变的染色体位置,紫色区域表示发生插入突变的位置,黄色区域表示发生重复突变的位置,而灰色线则表示发生片段位移的位置(fig.2f)。在鉴定到这些插入和缺失突变后,作者设计了特定引物验证了其中四个插入突变和一个位移突变,结果证明检测到的这些突变是准确的(fig.2e、fig.2g)。除了插入、缺失和位移突变外,作者还检测了基因重复突变。分析了整个染色体上重复突变的分布频率,并发现重复突变在端粒两端的位置出现频率非常高,这表明端粒处更容易发生基因重复事件(fig.2h)。
使用SMOOTH-seq检测K562细胞中的ecDNA
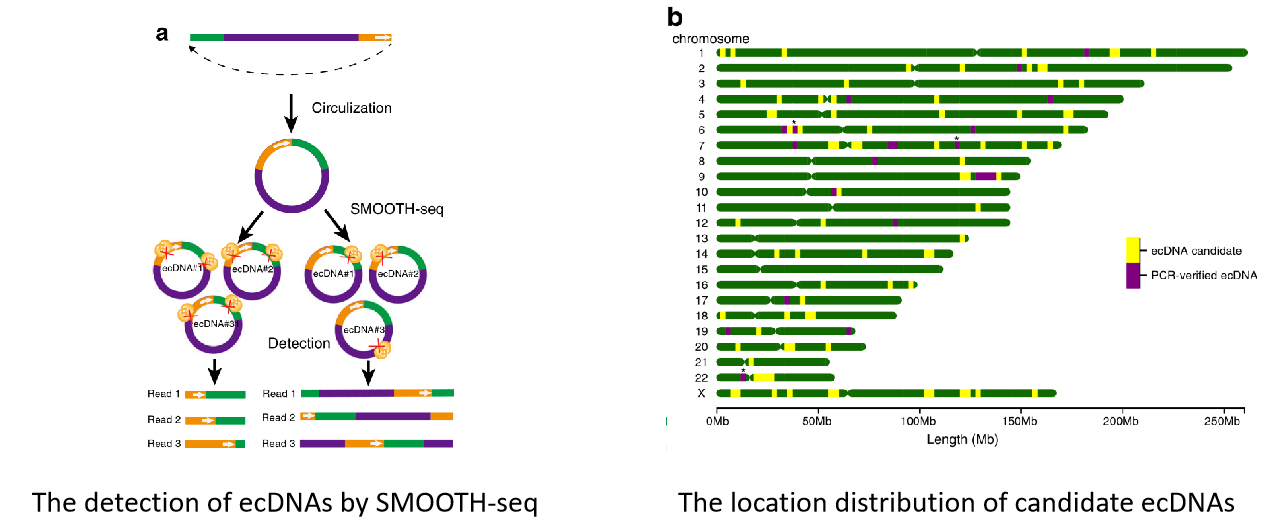
随后,作者尝试使用SMOOTH-seq检测染色体外的环状DNA(ecDNA)。ecDNA是一种特异于肿瘤的DNA结构,呈环状,大小约为1-4 Mb。它在癌细胞中普遍存在,而在正常细胞中很少检测到,这表明ecDNA是某些癌症的特征。图a展示了检测环状DNA的原理。当DNA片段的两端相连时,形成环状DNA。然后,tn转座酶结合带有接头序列的DNA,并从环状DNA中截取中间区域作为测序片段。当只有一个转座酶连接到环状DNA上时,整个环状DNA都会被检测到。作者最终鉴定到了共计125个候选ecDNA,并且这些ecDNA得到了至少两个K562细胞的测序数据的支持。在ecDNA区域内,共鉴定出了417个基因。这些基因在信号通路、免疫过程和细胞分裂等生物学方面的基因本体论中表现出富集现象(图b)。
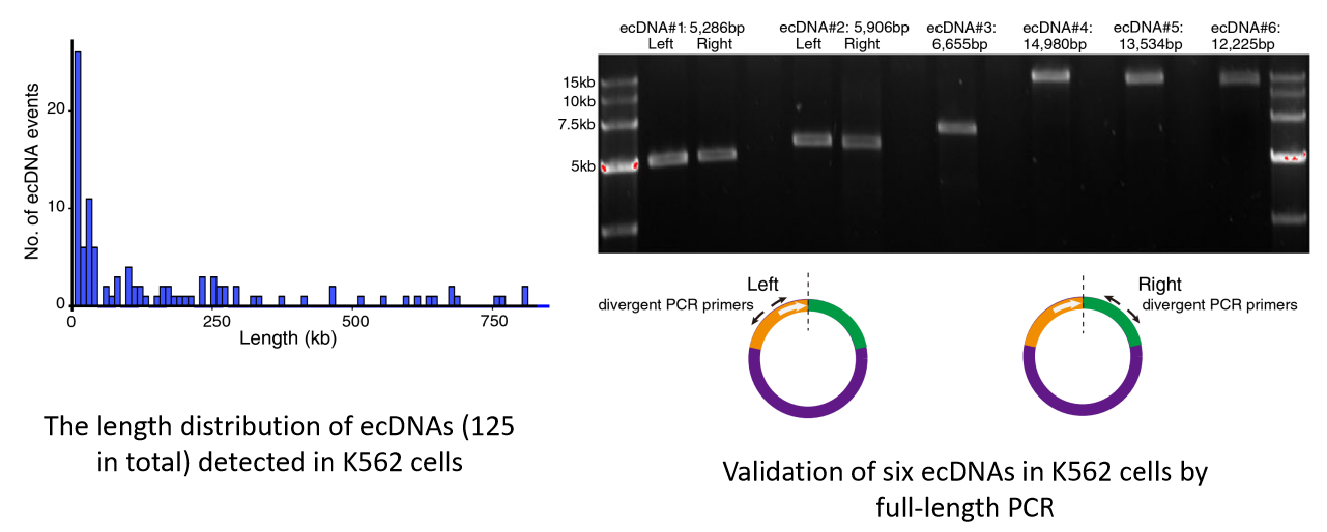
上图显示了检测到的所有环状DNA的长度分布,大多数环状DNA的长度在5Kb到1Mb之间。另外,作者选择了6个环状DNA分子进行PCR验证,并且使用了两种PCR方法进行相互验证,其中一种方法是设计上游引物在连接处进行扩增,另一种方法是设计下游引物在连接处进行扩增。如果两种PCR方法扩增出来的产物长度相同,那么就说明检测到的环状DNA是准确的。最终的结果表明,两种PCR扩增出的产物长度是相同的,验证了鉴定到的环状DNA的准确性。
标签:
bioinfo
北京 天气
多云
14℃


